|
|
[论文笔记] Attention U-Net: Learning Where to Look for the Pancreas
说在前面
因为是笔记,不是论文翻译,所以文字里还带有一些自己的思考,这篇文章的核心在于AGs,可以直接下跳Attention Gates for Image Analysis,但是为什么这么设计?还是需要阅读参考文献来做支撑。
发表于MIDL 18,原文地址:https://arxiv.org/abs/1804.03999。
本文作于2020年3月19日。
1、摘要
We propose a novel attention gate (AG) model for medical imaging that automatically learns to focus on target structures of varying shapes and sizes. 我们提出了一种应用于医学影像的基于attention gate的模型,它会自动学习区分目标的外形和尺寸。
Models trained with AGs implicitly learn to suppress irrelevant regions in an input image while highlighting salient features useful for a specific task. This enables us to eliminate the necessity of using explicit external tissue/organ localisation modules of cascaded convolutional neural networks (CNNs). 这种有attention gate的模型在训练时会学会抑制不相关的区域,注重有用的显著特征,这点对一个具体的任务来说很有效。这可以帮助我们不使用显示定位的组织和器官的CNN。
AGs can be easily integrated into standard CNN architectures such as the U-Net model with minimal compu- tational overhead while increasing the model sensitivity and prediction accuracy. Attention gate很容易被整合进标准的CNN模型中,极少的额外计算量却能带来显著的模型敏感度和准确率的提高。
The proposed Attention U-Net architecture is evaluated on two large CT abdominal datasets for multi-class image segmentation.Experimental results show that AGs consistently improve the prediction performance of U-Net across different datasets and training sizes while preserving computational efficiency. The source code for the proposed architecture is publicly available. 利用Attention U-Net模型,我们在两个大型CT腹部数据集上进行了多类别的图像分割。实验结果表明,AG可以在保持计算效率的同时,持续提高U-Net在不同数据集和训练规模下的预测性能。 代码开源,作者给出的PyTorch代码有多个2D/3D的版本。
2、前言
自动图像分割很重要,因为手动标记医学图像是一项琐碎且易错的事,所以需要准确的解决方案来提高临床工作的效率并辅助决策。
高表示能力、快速推理和卷积共享的特性使CNN成为事实上的图像分割标准,FCN和UNet是通用的架构。这类模型的特征表达能力很强,但是当目标器官在形状和大小方面表现出较大的患者间差异时,模型仍依赖于多级级联的CNN。级联框架提取感兴趣区域(ROI)并对该特定ROI进行密集的预测。(存疑,multi-stage cascaded CNNs该如何理解?) 但是,这种方法导致过多和冗余地使用计算资源和模型参数。 例如,级联中的所有模型都会重复提取相似的低级特征。
为了解决这个问题,文章提出了Attention gates (AGs)的方法,有以下几个优势:1、带AG的CNN可以直接端到端学习,无需额外的监督标注;2、在推理时会自动关注有显著特征的区域;3、不会引入大量的参数和计算量。AG通过抑制无关区域中特征的激活来提高模型灵敏度和准确性,以进行密集的标签预测。这样,可以在维持高准确率的同时消除使用外部器官定位模型的必要性。(这里针对将分割分为两步的方法,即检测+分割,先确定待分割的器官/组织的ROI,再进行小区域的分割。)
本文将Attention gates和U-Net结合(Attention U-Net)并应用于医学图像。我们选择具有挑战性的CT胰腺分割问题,为我们的方案做实验上的支撑。 由于组织对比度低以及器官形状和大小的可变性大,该任务有很大困难,同时根据两个常用的基准来评估:TCIA Pancreas CT-82和multi-class abdominal CT-150。 结果表明,AG可以在不使用多个CNN模型(multiple CNN models)的情况下实现state-of-the-art,在不同数据集和训练量范围内提高准确性。
CT胰腺分割:主要有statistical shape models、multi-atlas techniques和cascaded multi-stage CNN models三种方法。在CNN里,会使用初始粗粒度的模型(例如U-Net或回归森林)来获取ROI,然后使用裁剪后的ROI通过第二个模型进行细分。还有利用2D-FCN和递归神经网络(RNN)模型的组合来利用相邻轴向切片之间的依赖性。
Attention Gates:一般AG会被用在自然图像分析、知识图谱、图像描述、机器翻译和分类任务上。Attention机制最开始是通过解释输出类别分数相对于输入图像的梯度来探索注意力图。可训练的Attention机制根据设计可分为Hard attention和Soft attention。Hard attention主要是由迭代区域候选和区域裁剪,通常是不可微的,因此会使模型很难训练,通常依赖强化学习的其他参数;Soft attention是基于概率的,可以正常反向传播梯度。Channel-wise attention可以突出某些维度的特征,效果极好(SENet);Self-attention可以消除对外部信息(external gating information)的依赖。
在本文中,我们提出了一种新颖的self-attention gating模块,该模块可以在基于CNN的标准图像分析模型中用于密集标签预测。 此外,我们探索了AG对医学图像分析的好处,尤其是在图像分割的背景下。 这项工作的贡献可以总结如下:
- 提出基于网格的AG,这会使注意力系数更能凸显局部区域的特征。
- 首次在医学图像的CNN中使用Soft Attention,该模块可以替代分类任务中的Hard attention和器官定位任务中的定位模块。
- 将U-Net改进为Attention U-Net,增加了模型对前景像素的敏感度,并设计实验证明了这种改进是通用的。
3、具体方法
- Fully Convolutional Network
FCN已经成为医学图像的基准模型,因为鲁棒性和准确性远超传统方法。之所以有性能上的提升,(存疑)是因为以下三点:1.SGD优化;2.卷积核被所有像素共享;3.卷积操作很好地利用了图像的结构信息。(这里的FCN指全卷积网络这类网络,不是FCN-8s的那个模型)
CNN会根据一层一层的局部信息来提取高维图像的表示,最终在高维空间的离散像素会具有语义信息和巨大的感受野。基于U-Net构建的Attention U-Net,粗粒度的特征图会捕获上下文信息,并突出显示前景对象的类别和位置。随后,通过skip connections合并以多个比例提取的特征图,以合并粗粒度和细粒度的密集预测。
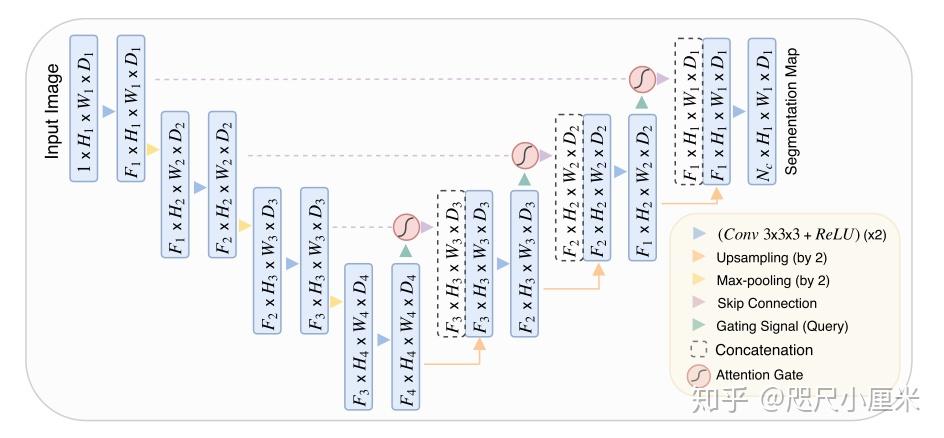
- Attention Gates for Image Analysis
CNN对形变程度大的小目标的false-positive(FP)预测是很困难,所以通常的做法先定位,然后再分割。CNN+AG也可以实现这个效果, 不需要训练多个模型和大量额外的参数,AG会抑制了无关背景区域中的特征响应,而无需在网络之间裁剪ROI。
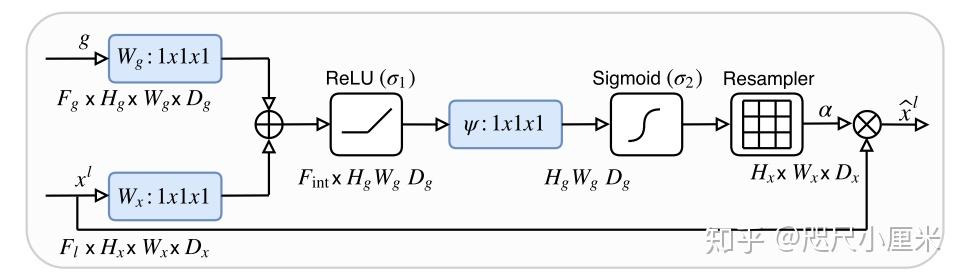
Attention coefficient( \alpha_i\in(0,1] )是为了突出显著的图像区域和抑制任务无关的特征响应, \alpha_i 和feature map的乘法是Element-wise的(对应元素逐个相乘)。如果存在多个语义类别,建议学习多维注意力系数。门控向量包含上下文信息,以修剪低级特征响应。论文中选择加性注意力来获得门控系数,尽管这在计算上更昂贵(加法要比乘法更容易吧?),但从实验上看,它的性能比乘法注意力要高。
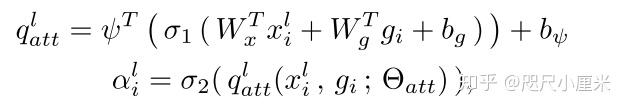
上图为加性注意力的公式,注意这里要结合结构来一起分析, \sigma_1 是Relu函数, \sigma_2 是Sigmoid函数, W_g、W_x、\psi 都是卷积操作, b_g、b_\psi 都是对应卷积的偏置项(所以发现 W_x 无偏置)。 F_{int} 一般比 F_g、F_l 要小。
在图像标注和分类任务中一般都用Softmax函数,之所以 \sigma_2 使用Sigmoid函数,是因为顺序使用softmax函数会输出较稀疏的激活响应,而用Sigmoid函数能够使训练更好的收敛。门控信号不是全局图像的表示矢量,而是在一定条件下部分图像空间信息的grid signal,每个skip connection的门控信号都会汇总来自多个成像比例的信息。(值得思考)
有一个细节我觉得值得思考,就是这个最后的Resampler是做什么的?在官方的源码中是做上采样的,但是AGs最开始 1*1 卷积之后做了相加,这意味2个输出的尺寸应该是一致的,那么什么情况下需要上采样呢?若是2个输入最开始尺寸不一致,那又是如何按位相加的呢?根据论文的结构图,2个输入的尺寸是一样的,所以这个Resampler得留坑了。
the first skip connections, are not used in the gating function since they do not represent the input data in a high dimensional space.
这里提到第一个上采样不需要skip connection,但是结构图里还是做了,迷。
- Attention Gates in U-Net Model
这里推导了一下如何反向传播更新的。
部分对AGs的解释:为了减少可训练参数的数量和AG的计算复杂性,执行线性变换时无需任何空间支持( 1*1*1 卷积),并且将输入的feature map下采样到门控信号的维度(降维),相应的线性变换将特征图解耦,并将其映射到较低维空间以进行门操作。模型会强制中间特征图在每个图像尺度的语义上具有区别性,这有助于确保不同尺度上的注意力单元具有影响对大范围前景内容的响应的能力。
4、实验和结果
AGs是模块化的,与应用类型无关; 因此,它可以很容易地适应分类和回归任务。为了证明其对图像分割的适用性,我们在具有挑战性的腹部CT多标签分割问题上评估Attention U-Net模型。特别是,由于形状变化和组织对比度差,胰腺边界描绘是一项艰巨的任务。 我们的模型在分割性能,模型容量,计算时间和内存要求方面与标准3D U-Net进行了比较。
- 评估数据集:NIH-TCIA 和 这篇论文中的。
- 实施细节:有一个3D的模型,Adam,BN,deep-supervision(留坑思考)和标准数据增强技术(仿射变换,轴向翻转,随机裁剪)。
- 注意力图分析:我们通常观察到AG最初具有均匀分布并且在所有位置,然后逐步更新和定位到目标器官边界。在较粗糙的尺度上,AG提供了粗略的器官轮廓,这些器官在更精细的分辨率下逐渐细化。 此外,通过在每个图像尺度上训练多个AG,我们观察到每个AG学习专注于器官的特定子集。
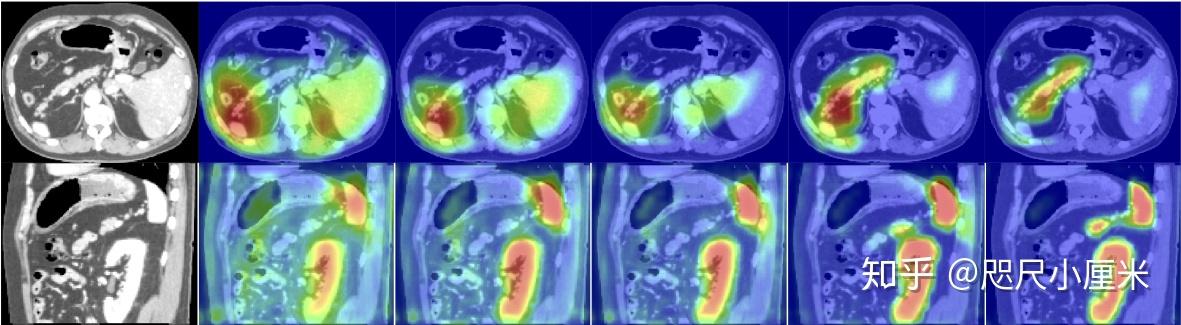
5、讨论和总结
尽管我们的残差连接实验并未提供任何显着的性能改进,但未来的研究将集中在此方面,以获得更好的训练性能。最后,我们注意到随着改进的GPU计算能力和内存的出现,可以以更大的批处理量训练容量更大的3D模型,而无需进行图像下采样。 这样,我们就不需要利用临时的后处理技术来进一步改善最新的结果。 同样,可以通过使用高分辨率输入批处理来进一步提高Attention U-Net的性能,而无需使用其他试探法。
6、代码
2D版本。因为可能会爆显存,所以加了scale_factor来调整卷积核的数量。
import numpy as np
import torch
import torch.nn as nn
class Attention_block(nn.Module):
def __init__(self, F_g, F_l, F_int):
super(Attention_block, self).__init__()
self.W_g = nn.Sequential(
nn.Conv2d(F_g, F_int, kernel_size=1, stride=1, padding=0, bias=True),
nn.BatchNorm2d(F_int)
)
self.W_x = nn.Sequential(
nn.Conv2d(F_l, F_int, kernel_size=1, stride=1, padding=0, bias=True),
nn.BatchNorm2d(F_int)
)
self.psi = nn.Sequential(
nn.Conv2d(F_int, 1, kernel_size=1, stride=1, padding=0, bias=True),
nn.BatchNorm2d(1),
nn.Sigmoid()
)
self.relu = nn.ReLU(inplace=True)
def forward(self, g, x):
g1 = self.W_g(g)
x1 = self.W_x(x)
psi = self.relu(g1 + x1)
psi = self.psi(psi)
return x * psi
class conv_block(nn.Module):
def __init__(self, ch_in, ch_out):
super(conv_block, self).__init__()
self.conv = nn.Sequential(
nn.Conv2d(ch_in, ch_out, kernel_size=3, stride=1, padding=1, bias=True),
nn.BatchNorm2d(ch_out),
nn.ReLU(inplace=True),
nn.Conv2d(ch_out, ch_out, kernel_size=3, stride=1, padding=1, bias=True),
nn.BatchNorm2d(ch_out),
nn.ReLU(inplace=True)
)
def forward(self, x):
return self.conv(x)
class up_conv(nn.Module):
def __init__(self, ch_in, ch_out):
super(up_conv, self).__init__()
self.up = nn.Sequential(
nn.Upsample(scale_factor=2),
nn.Conv2d(ch_in, ch_out, kernel_size=3, stride=1, padding=1, bias=True),
nn.BatchNorm2d(ch_out),
nn.ReLU(inplace=True)
)
def forward(self, x):
return self.up(x)
class AttU_Net(nn.Module):
def __init__(self, n_channels=3, n_classes=1, scale_factor=1):
super(AttU_Net, self).__init__()
filters = np.array([64, 128, 256, 512, 1024])
filters = filters // scale_factor
self.n_channels = n_channels
self.n_classes = n_classes
self.scale_factor = scale_factor
self.Maxpool = nn.MaxPool2d(kernel_size=2, stride=2)
self.Conv1 = conv_block(ch_in=n_channels, ch_out=filters[0])
self.Conv2 = conv_block(ch_in=filters[0], ch_out=filters[1])
self.Conv3 = conv_block(ch_in=filters[1], ch_out=filters[2])
self.Conv4 = conv_block(ch_in=filters[2], ch_out=filters[3])
self.Conv5 = conv_block(ch_in=filters[3], ch_out=filters[4])
self.Up5 = up_conv(ch_in=filters[4], ch_out=filters[3])
self.Att5 = Attention_block(F_g=filters[3], F_l=filters[3], F_int=filters[2])
self.Up_conv5 = conv_block(ch_in=filters[4], ch_out=filters[3])
self.Up4 = up_conv(ch_in=filters[3], ch_out=filters[2])
self.Att4 = Attention_block(F_g=filters[2], F_l=filters[2], F_int=filters[1])
self.Up_conv4 = conv_block(ch_in=filters[3], ch_out=filters[2])
self.Up3 = up_conv(ch_in=filters[2], ch_out=filters[1])
self.Att3 = Attention_block(F_g=filters[1], F_l=filters[1], F_int=filters[0])
self.Up_conv3 = conv_block(ch_in=filters[2], ch_out=filters[1])
self.Up2 = up_conv(ch_in=filters[1], ch_out=filters[0])
self.Att2 = Attention_block(F_g=filters[0], F_l=filters[0], F_int=filters[0] // 2)
self.Up_conv2 = conv_block(ch_in=filters[1], ch_out=filters[0])
self.Conv_1x1 = nn.Conv2d(filters[0], n_classes, kernel_size=1, stride=1, padding=0)
def forward(self, x):
# encoding path
x1 = self.Conv1(x)
x2 = self.Maxpool(x1)
x2 = self.Conv2(x2)
x3 = self.Maxpool(x2)
x3 = self.Conv3(x3)
x4 = self.Maxpool(x3)
x4 = self.Conv4(x4)
x5 = self.Maxpool(x4)
x5 = self.Conv5(x5)
# decoding + concat path
d5 = self.Up5(x5)
x4 = self.Att5(g=d5, x=x4)
d5 = torch.cat((x4, d5), dim=1)
d5 = self.Up_conv5(d5)
d4 = self.Up4(d5)
x3 = self.Att4(g=d4, x=x3)
d4 = torch.cat((x3, d4), dim=1)
d4 = self.Up_conv4(d4)
d3 = self.Up3(d4)
x2 = self.Att3(g=d3, x=x2)
d3 = torch.cat((x2, d3), dim=1)
d3 = self.Up_conv3(d3)
d2 = self.Up2(d3)
x1 = self.Att2(g=d2, x=x1)
d2 = torch.cat((x1, d2), dim=1)
d2 = self.Up_conv2(d2)
d1 = self.Conv_1x1(d2)
return d1 |
|